Rでカイ二乗分布の確率密度関数のグラフをシミュレーション的に描く
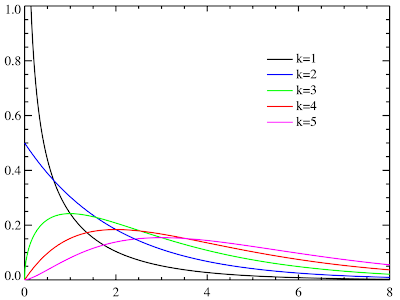
カイ二乗分布といえば、独立性の検定や適合度の検定で使ったりしますよね。 ウィキペディア には、確率密度関数のグラフが載っていて↓こんな感じ。 カイ二乗分布の確率密度関数のグラフ このグラフをRで描いてみようと、確率密度関数の式を見てみると、 ↑こんなのが載っていて、なんだかすごくむつかしい。Γ(ガンマ)関数ってのが出てきて、さらにそれは積分の形で定義されていたりして。 でもまあ、そのへんは理解できなくても、Rにはカイ二乗分布の確率密度関数(dchisq)が用意されているので、下記のようにすれば、ウィキペディアに載っていたのとそっくりのグラフが描けます。 curve ( dchisq ( x , 1 ) , xlim= c ( 0 , 8 ) , ylim= c ( 0 , 1 ) , col = "black" , ylab= "dchisq(x, k)" ) curve ( dchisq ( x , 2 ) , xlim= c ( 0 , 8 ) , ylim= c ( 0 , 1 ) , col = "blue" , add=T ) curve ( dchisq ( x , 3 ) , xlim= c ( 0 , 8 ) , ylim= c ( 0 , 1 ) , col = "green" , add=T ) curve ( dchisq ( x , 4 ) , xlim= c ( 0 , 8 ) , ylim= c ( 0 , 1 ) , col = "red" , add=T ) curve ( dchisq ( x , 5 ) , xlim= c ( 0 , 8 ) , ylim= c ( 0 , 1 ) , col = "magenta" , add=T ) #凡例 legend ( "topright" , lty= 1 , legend = c ( "k=1" , "k=2" , "k=3" , &qu